腸内細菌の新規な乳酸応答機構を同定 ~腸内環境の理解や乳酸菌を含む食品への応用に期待~
2023年06月02日
明治大学
腸内細菌の新規な乳酸応答機構を同定
~腸内環境の理解や乳酸菌を含む食品への応用に期待~
明治大学農学部農芸化学科 ゲノム微生物研究室の島田 友裕 准教授と来島 楓(博士前期課程2年)、中元 颯馬(博士前期課程1年)、安西 拓実(2021年博士前期課程修了)、藤森 美希(2019年農学部卒業)は、大腸菌の乳酸応答転写因子LldRが、乳酸を炭素源として利用する遺伝子群や、乳酸の酸ストレスに適応するための遺伝子群を活性化することを明らかにしました。本研究成果により、腸内細菌が乳酸に適応するための新たな分子機構が明らかとなり、乳酸菌を含む食品や飲料の効果の理解・応用が期待されます。
発酵乳や乳酸菌飲料に含まれる乳酸菌は、腸内で多量の乳酸を作ります。この効果によって有害菌の増殖が抑えられ、腸内腐敗の防止、腸内菌叢の正常化につながります。しかしながら、腸内細菌の乳酸に応答する分子機構は良く分かっておりませんでした。本学研究室では、大腸菌をモデル微生物として、大腸菌が持つ全ての転写制御因子の機能解明を目指しています。その一環で本研究では、乳酸に応答する転写因子LldRのゲノム制御ネットワークの解析を行いました。その結果、LldRは乳酸輸送体や乳酸デヒドロゲナーゼといった乳酸を炭素源として利用する遺伝子だけでなく、グルタミン酸を用いた酸耐性、膜脂肪酸組成の変化、グリコール酸の利用など、様々な機能の遺伝子群を活性化していることが分かりました。さらに、乳酸菌と共培養させた際の大腸菌の生存率にも、LldRが大きな影響を及ぼすことを実証しました。本研究成果により、腸内細菌が乳酸に応答するための仕組みが分子レベルで明らかになりました。 本研究は、日本学術振興会科学研究費補助金、ロッテ財団研究助成、住友財団研究助成の支援を受けました。研究成果は原著論文として、英国の国際学術誌「Microbial Genomics」(電子版)に2023年5月23日付で掲載されました。(DOI: 10.1099/mgen.0.001015.)
生物はゲノムに持つ遺伝子を選択的に利用することで環境に適応しており、その仕組みを理解することは、ポストゲノム時代の生命科学分野における先端的研究課題の一つです。モデル微生物である大腸菌はゲノムに約4700の遺伝子を持っており、それらは約300種類の転写制御因子により制御されていることが分かってきていることから、それら全転写制御因子の機能解明が課題となっています。本研究の解析対象である転写因子LldRは、これまでに乳酸に結合し活性型となり、乳酸代謝遺伝子群を活性化することで、炭素源としての乳酸の利用を制御していることが分かっていましたが、ゲノム全体における役割は不明でした。
島田准教授らは大腸菌をモデル微生物として、一つの生物のゲノム転写制御機構の全体像の理解を目指しています。その一環で、LldRについて、Genomic SELEX(gSELEX)法を用いてゲノム上の結合領域を解析したところ、既知標的遺伝子群を含む約20個の遺伝子群を標的としていることが分かりました(図1)。新規の標的遺伝子群には、グルタミン酸を用いた酸耐性、膜脂肪酸組成の変化、グリコール酸の代謝などに関与する遺伝子群が含まれていました。さらなる解析により、LldRによりこれらの遺伝子群が活性化されていることや(図2)、乳酸の酸ストレスに耐性を獲得していることが分かりました(図3)。さらに、大腸菌と乳酸菌の共培養実験では、乳酸菌の生産する乳酸の酸ストレス(pHの低下)に対し、LldRが重要な役割を持つことを実証しました(図4)。なお、FlhDCは腸内細菌種間で広く保存されています。これらのことから、腸内細菌は環境中の乳酸濃度の変化に乳酸応答転写因子LldRにより応答することで、乳酸を炭素源として利用したり、乳酸の酸としてのストレスに適応したりしていることが示唆されました(図5)。
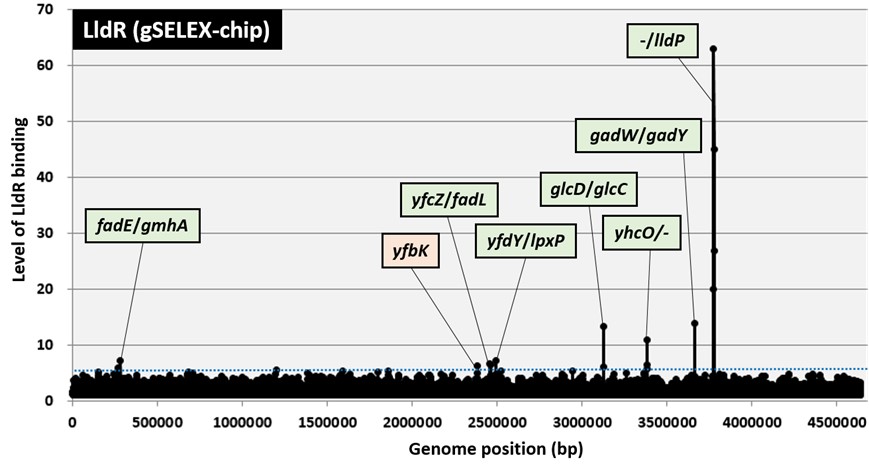
図1.本研究グループにより独自に開発されたGenomic SELEX法を用いて同定された乳酸応答転写因子LldRの大腸菌ゲノム上の結合領域。横軸が大腸菌K-12株のゲノム領域、縦軸がLldRの結合強度を示す。結合領域に隣接する遺伝子名が示されている(緑色は遺伝子間領域、橙色は遺伝子上領域を示す)。
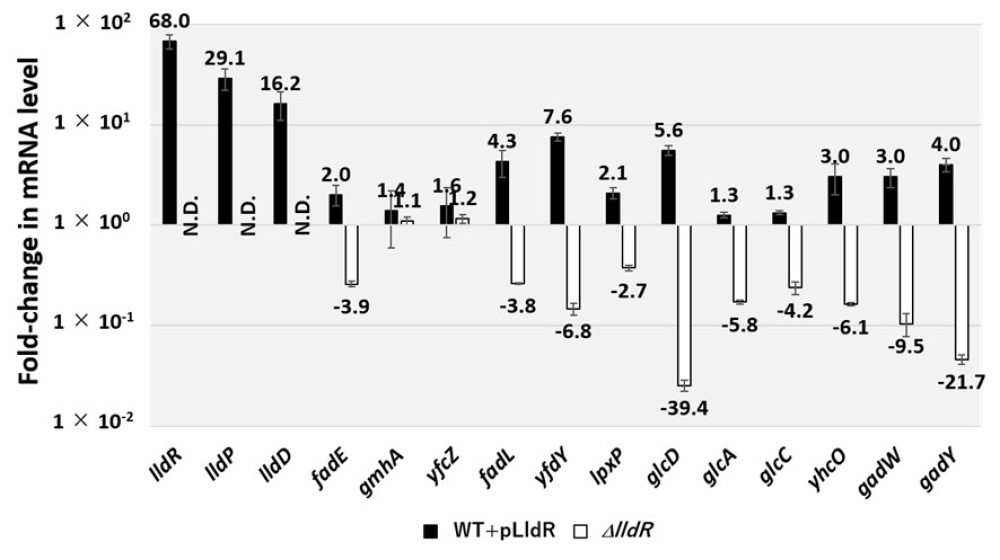
図2.大腸菌野生株とlldR欠損株、LldR過剰発現株における標的遺伝子群のmRNAレベルの比較解析。
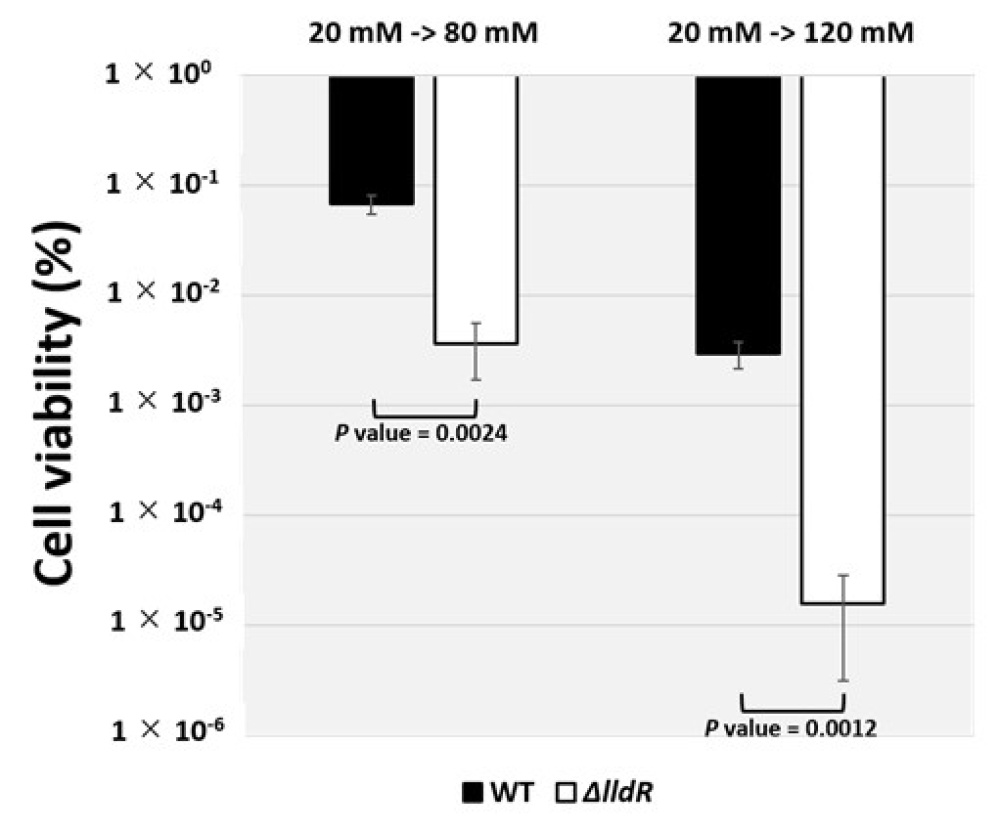
図3.高濃度乳酸添加時における大腸菌野生株とlldR欠損株の生存率の観察結果。
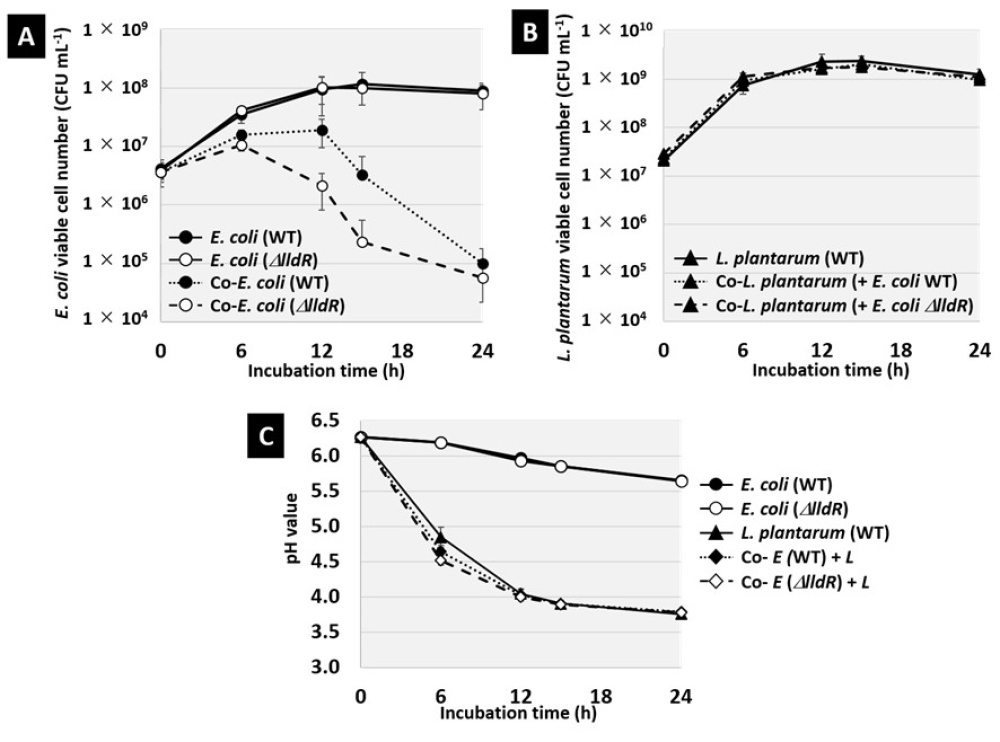
図4.単菌培養および乳酸菌との共培養の際の大腸菌野生株およびlldR欠損株の生菌数と培地中pHの経時的観察結果。Aが大腸菌の生菌数、Bが乳酸菌の生菌数、Cが培地中のpHの変化。
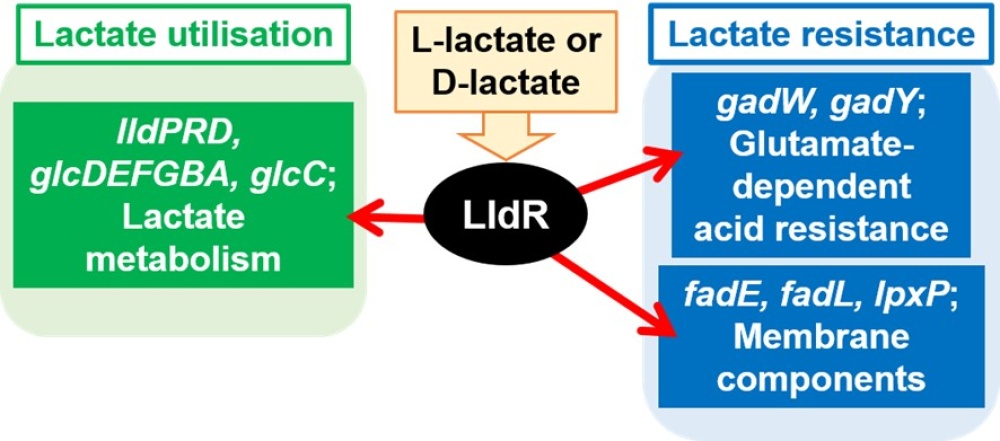
図5.本研究成果から得られた乳酸応答機構のモデル図。腸内細菌は環境中の乳酸濃度の変化に乳酸応答転写因子LldRにより応答することで、乳酸を炭素源として利用したり、乳酸の酸としてのストレスに適応したりしている。
要旨
研究成果のポイント
- 大腸菌の乳酸応答転写因子LldRのゲノム上結合領域および標的遺伝子群の網羅的な同定に成功した。既知標的の乳酸の代謝遺伝子群に加え、新規標的遺伝子群には、グルタミン酸を用いた酸耐性、膜脂肪酸組成の変化、グリコール酸の代謝などが含まれていた。
- LldRが実際にこれらの遺伝子群を活性化することが分かった。さらに、LldRにより、低濃度の乳酸存在下(乳酸の炭素源としての利用)では生育が促進されること、高濃度の乳酸存在下(乳酸の酸ストレスとしての耐性)では生存率が上昇すること、さらに、乳酸菌との共培養においても生存率が上昇することを実証した。
- 乳酸および乳酸菌に対する腸内細菌の応答機構が、分子レベルで実証された。本研究成果は、乳酸菌を含む食品や飲料の効果の理解・応用に役立つ。
1.研究の背景
2.研究内容と成果
3.今後の期待
本研究グループは一つの生物の遺伝子発現制御機構の全体像を理解する目的で、大腸菌をモデル生物としてこれまでに数々の転写制御因子の機能同定に成功してきました。特に、試験管内でゲノム上の直接的な結合配列を網羅的に同定するために独自に開発したgSELEX法を用いた研究戦略により、これまでに大腸菌K-12株の持つ7種類のRNAポリメラーゼシグマ因子のうち6種類、約300種類の転写因子のうち70種類以上の機能同定に成功してきました。本研究で解析したLldRは乳酸代謝に関わる遺伝子群を制御していることが報告されていましたが、ゲノム全体の直接的な制御を改めて解析したことにより、酸ストレス耐性や膜脂肪酸組成といった酸ストレスとしての乳酸に適応するための遺伝子群も制御していることが分かりました。今後も、本研究グループによるgSELEX法を用いた転写制御因子の機能解析により、微生物のゲノムを利用する仕組みの本質が明らかとなり、微生物の新たな生存戦略や仕組みが明らかとなることが期待されます。
発酵乳や乳酸菌飲料に含まれる乳酸菌の乳酸を作る効果によって、有害菌の増殖を抑えることが、腸内腐敗の防止、腸内菌叢の正常化につながっています。本研究成果は、発酵乳や乳酸菌飲料の腸内細菌に対する効果の理解や応用にも役立つことが期待されます。
4.発表論文
〈タイトル〉
Expanded roles of lactate-sensing LldR in transcription regulation of the Escherichia coli K-12 genome: lactate utilization and acid resistance.
〈著者名〉
Takumi Anzai, Kaede Kijima, Miki Fujimori, Soma Nakamoto, Akira Ishihama and Tomohiro Shimada
〈雑誌名〉
Microbial Genomics
〈DOI〉
【研究グループ】
明治大学 農学部農芸化学科 ゲノム微生物学研究室
准教授 島田 友裕(しまだ ともひろ)
農学研究科博士前期課程2年 来島 楓(きじま かえで)
農学研究科博士前期課程1年 中元 颯馬(なかもと そうま)
2021年農学研究科博士前期課程修了生 安西 拓実(あんざい たくみ)
2019年農学部農芸化学科卒業生 藤森 美希(ふじもり みき)
参考図

図1.本研究グループにより独自に開発されたGenomic SELEX法を用いて同定された乳酸応答転写因子LldRの大腸菌ゲノム上の結合領域。横軸が大腸菌K-12株のゲノム領域、縦軸がLldRの結合強度を示す。結合領域に隣接する遺伝子名が示されている(緑色は遺伝子間領域、橙色は遺伝子上領域を示す)。

図2.大腸菌野生株とlldR欠損株、LldR過剰発現株における標的遺伝子群のmRNAレベルの比較解析。

図3.高濃度乳酸添加時における大腸菌野生株とlldR欠損株の生存率の観察結果。

図4.単菌培養および乳酸菌との共培養の際の大腸菌野生株およびlldR欠損株の生菌数と培地中pHの経時的観察結果。Aが大腸菌の生菌数、Bが乳酸菌の生菌数、Cが培地中のpHの変化。

図5.本研究成果から得られた乳酸応答機構のモデル図。腸内細菌は環境中の乳酸濃度の変化に乳酸応答転写因子LldRにより応答することで、乳酸を炭素源として利用したり、乳酸の酸としてのストレスに適応したりしている。
- お問い合わせ先
-
内容に関するお問い合わせ
明治大学農学部農芸化学科 ゲノム微生物学研究室 准教授 島田 友裕
TEL: 044-934-7102
E-mail: tomoshimada@meiji.ac.jp -
取材に関するお問い合わせ
取材お申し込みフォームから必要事項をご記入のうえ、送信してください。
取材お申し込みフォーム
問題なく送信された場合、お申し込み完了をお知らせするメールが自動送信されますのでご確認ください。
お急ぎの場合は、電話でもご連絡ください。
明治大学 経営企画部 広報課
TEL:03-3296-4082
MAIL:koho@mics.meiji.ac.jp


