明治大学 農学部 農芸化学科 島田友裕専任講師ら 大腸菌の転写制御ネットワークの全体像を解明
2018年03月16日
明治大学
明治大学 農学部 農芸化学科 島田友裕専任講師ら
大腸菌の転写制御ネットワークの全体像を解明
大腸菌の転写制御ネットワークの全体像を解明
明治大学農学部 農芸化学科 応用生化学研究室(島田友裕専任講師)らが、大腸菌の転写制御ネットワークの全体像の解明に関する論文を、Oxford academicのNucleic Acids Research誌に発表しました。
・本研究グループは、細胞の転写制御ネットワークの全体像を理解するために、独自に開発したGenomic SELEX法を用いて、大腸菌の持つ約300種類の転写因子のうち、200種類以上のゲノム上の結合領域を同定してきました。
・転写因子のゲノム上の結合領域の特徴により、転写因子の新分類を提案しました。さらに、ゲノム上の結合領域が一箇所であるSingle-target regulatorsに着目し、その特徴を明らかとしました。
・本研究は、ひとつの生物の全ての転写因子および全ての遺伝子を対象にしており、その成果は、生物が遺伝子を利用する仕組みの全体像を理解するための重要な知見となります。
近年ではゲノム配列を決定する事は容易になりつつあり、生物の持つ遺伝子数やその機能の全体像の理解が可能となってきました。ゲノムにある遺伝子のどれが利用されるかは、DNAを鋳型にRNAが合成される転写の段階で調節され、転写装置であるRNAポリメラーゼおよび環境変化を感知する転写因子の相互作用によって制御されています。これまで、遺伝子個別の転写制御に関しては、細菌からヒトにいたるまで盛んに研究が行われ、関与する転写因子とその作用機序が同定されてきました。しかしながら、遺伝子を利用する仕組みの全体像については、異なる生物における研究成果や情報を寄せ集めて推測しているのが現状です。そのため、ゲノムの全ての遺伝子を対象にした生物丸ごとの転写制御ネットワークの解明には程遠く、新たな研究戦略が求められていました。
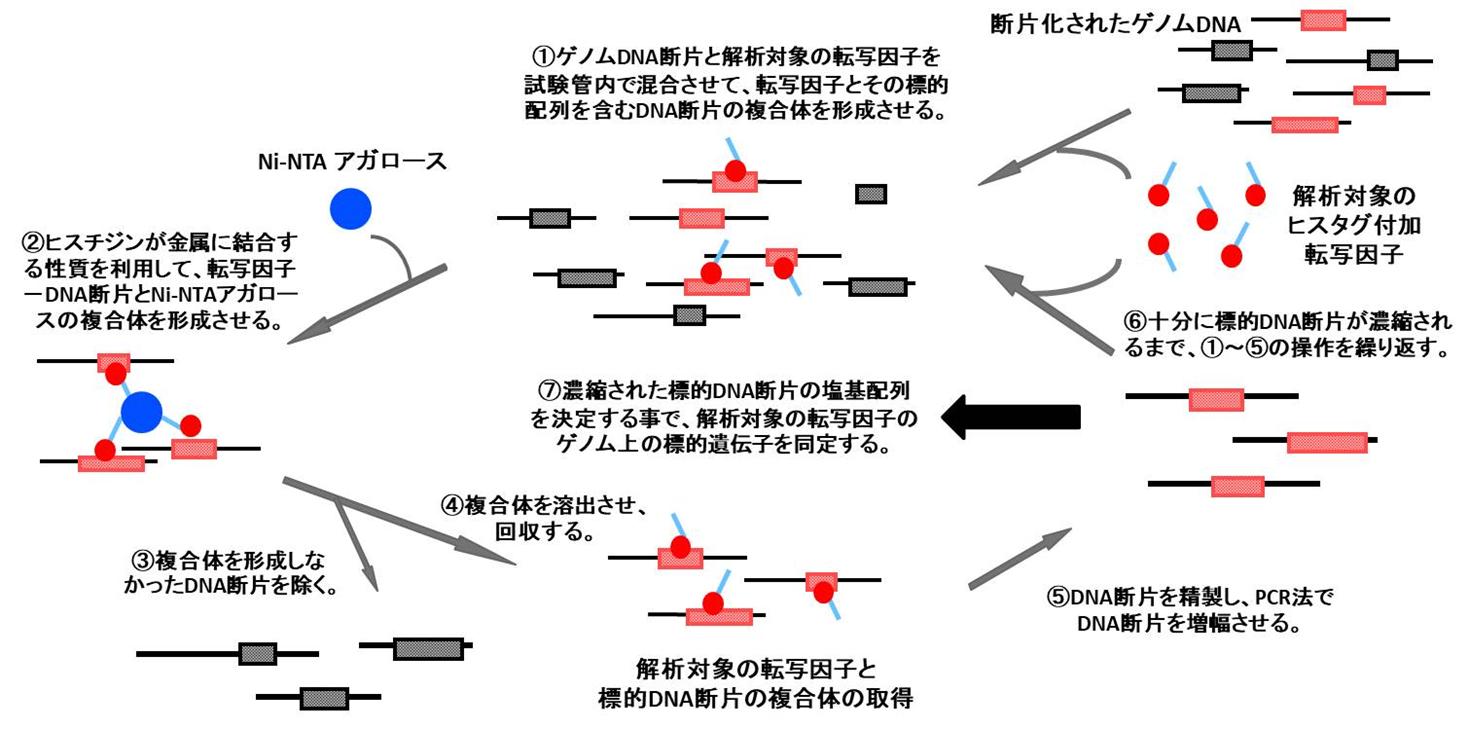
大腸菌のゲノムには、遺伝子が約4,500個しかなく、個々の遺伝子の機能が最も解析されているモデル生物です。そのゲノムの転写制御は、約300種類の転写因子とゲノムDNAとの相互作用によって行われています。転写因子の役割によって制御する遺伝子は異なり、ゲノム上の結合領域も異なります。本研究グループでは、そのうちの200種類以上の転写因子を、新規に開発したGenomic SELEX法(図1)を用いてゲノム上の結合領域を決定し、その情報からそれぞれの転写因子が支配する直接的な標的遺伝子群を明らかにしました。転写因子はゲノム上で拮抗阻害し合う事が判明しており、他因子存在下における解析では全標的遺伝子の同定が困難となっていますが、本研究で用いたGenomic SELEX法では、他因子の非存在下におけるゲノム上の全標的遺伝子を同定する事が可能です。
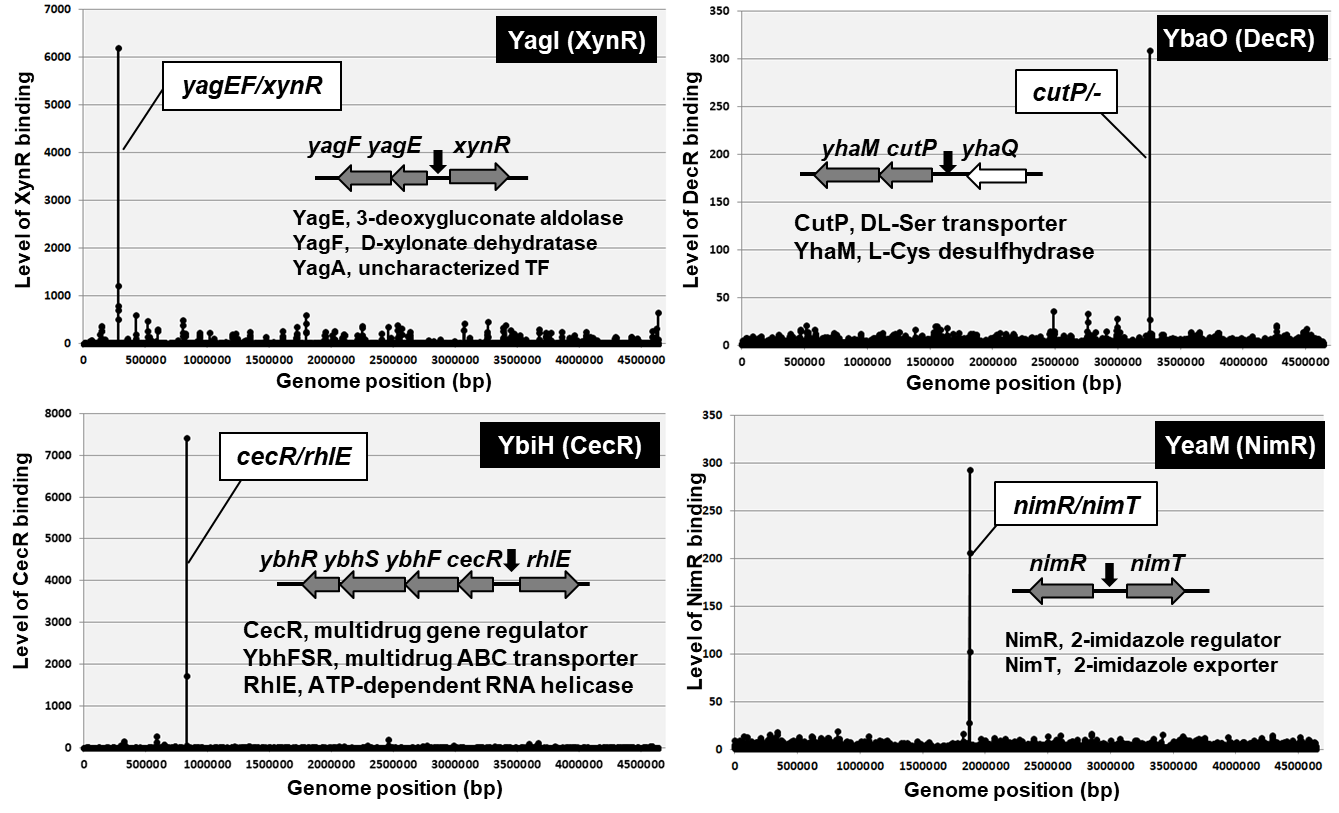
今回、本研究グループの研究成果を基盤として、転写因子の結合領域数および機能を詳細に解析しました。そして、ゲノム上の結合領域数が千箇所前後の転写因子をNucleoid-associated regulators、数百箇所をGlobal regulators、数十箇所をLocal regulators、一箇所であるものをSingle-target regulatorsとして分類する事を提案しました(図2)。さらに、Single-target regulatorsについて、その機能の特徴により、単独結合型、エフェクター存在下活性化型、リン酸化活性化型に小分類する事を提案しました(図3)。また、新たに複数のSingle-target regulatorsを同定しました(図4)。これらの成果から、大腸菌の転写制御ネットワークの全体像を提案し、Oxford AcademicのNucleic Acids Research誌上で発表しました。
これまでにも大腸菌で働く転写因子とその標的遺伝子は、様々な研究グループが明らかにしようとしてきましたが、実験に用いる大腸菌標準株でさえも遺伝的背景が異なる事が判明しており、異なる研究室からの論文データの寄せ集めでは、全体像の正しい理解は得られませんでした。一方、本研究では遺伝的背景が等しい単一菌株を利用し、ひとつの研究室で実施された研究成果である事が特徴です。本研究は、ひとつの生物で働く全転写因子の制御機能解明、および、生物が遺伝子を利用する仕組みの全体像の理解への先駆けとなる事が期待されます。
Single-target regulators form a minor group of transcription factors in Escherichia coli K-12.
Tomohiro Shimada, Hiroshi Ogasawara, Akira Ishihama
Nucleic Acids Research.
2018年2月26日 (https://doi.org/10.1093/nar/gky138)
明治大学 農学部農芸化学科 専任講師 島田友裕
信州大学 基盤研究支援センター 助教 小笠原寛
法政大学 マイクロ・ナノテクノロジー研究センター 特任教授 石浜明


本研究成果のポイント
・転写因子のゲノム上の結合領域の特徴により、転写因子の新分類を提案しました。さらに、ゲノム上の結合領域が一箇所であるSingle-target regulatorsに着目し、その特徴を明らかとしました。
・本研究は、ひとつの生物の全ての転写因子および全ての遺伝子を対象にしており、その成果は、生物が遺伝子を利用する仕組みの全体像を理解するための重要な知見となります。
背景
研究手法と成果
今回、本研究グループの研究成果を基盤として、転写因子の結合領域数および機能を詳細に解析しました。そして、ゲノム上の結合領域数が千箇所前後の転写因子をNucleoid-associated regulators、数百箇所をGlobal regulators、数十箇所をLocal regulators、一箇所であるものをSingle-target regulatorsとして分類する事を提案しました(図2)。さらに、Single-target regulatorsについて、その機能の特徴により、単独結合型、エフェクター存在下活性化型、リン酸化活性化型に小分類する事を提案しました(図3)。また、新たに複数のSingle-target regulatorsを同定しました(図4)。これらの成果から、大腸菌の転写制御ネットワークの全体像を提案し、Oxford AcademicのNucleic Acids Research誌上で発表しました。
今後の期待
発表論文
タイトル
著者名
雑誌名
オンライン発表
研究グループ
信州大学 基盤研究支援センター 助教 小笠原寛
法政大学 マイクロ・ナノテクノロジー研究センター 特任教授 石浜明
【図1】本研究グループにより独自に開発されたGenomic SELEX法。

【図2】本研究により提案されたゲノム上の標的数による転写因子の新分類。大腸菌ゲノムにある約4,500個の遺伝子と、それを制御する約300種類の転写因子との対応関係が見て取れます。一つの生物丸ごとの遺伝子発現を制御する仕組みを理解するための重要な知見となります。

【図3】ゲノム上の結合領域が1つであるSingle-target regulatorsの新規機能分類。転写因子が活性化する(DNA結合型になる)ために必要な条件によって分類しました。転写因子単独で活性化しており、エフェクター(化合物)存在下で不活性化するものをGroup 1、転写因子単独では不活性化しており、エフェクターが存在する条件下で活性化するものをGroup 2、転写因子がリン酸化される事により活性化するものをGroup 3としました。

【図4】本研究グループにより、新規に機能が同定されたSingle-target regulatorsの4例。横軸に大腸菌ゲノムの位置、縦軸に転写因子の結合強度を示しました。いずれの転写因子もゲノム上で一箇所の強い結合が検出されました。近年まで機能未知であった転写因子ですが、YagIはキシロン酸代謝、YbaOはシステインの解毒、YbiHはセフォペラゾン・クロラムフェニコール耐性、YeaMは2-ニトロイミダゾール耐性の遺伝子を制御している事を同定し、それぞれ、XynR (Regulator of xylonate catabolism), DecR (Regulator of detoxification of cysteine), CecR (Regulator of cefoperazone and chloramphenicol sensitivity), NimR (Regulator of nitroimidazole transporter)と命名する事を提案しました。





- お問い合わせ先
-
研究内容に関するお問い合わせ先
明治大学 農学部 農芸化学科 応用生化学研究室(島田友裕専任講師)
TEL: 044-934-7102Email:tomoshimada@meiji.ac.jp
-
取材に関するお問い合わせ先
明治大学 広報課
TEL: 03-3296-4330Email:koho@mics.meiji.ac.jp


