微生物の定常期ストレス応答のための新規転写因子を同定~微生物の自然環境下における生存の理解・応用に期待~
2022年06月09日
明治大学
微生物の定常期ストレス応答のための新規転写因子を同定
~微生物の自然環境下における生存の理解・応用に期待~
~微生物の自然環境下における生存の理解・応用に期待~
明治大学農学部農芸化学科ゲノム微生物学研究室の島田 友裕准教授、小林 一幾博士研究員は、法政大学マイクロ・ナノテクノロジー研究センターの石浜 明客員教授の研究グループ、および日本電信電話株式会社(NTT)宇宙環境エネルギー研究所の今村 壮輔特別研究員、高谷 和宏主幹研究員との共同研究により、大腸菌の定常期ストレス応答に関わる新規の転写因子を同定してその機能を解明しました。本研究成果は、微生物が自然環境下に適応するための仕組みの理解・応用に役立ちます。
生物はゲノムに持つ遺伝子を選択的に利用することで環境に適応しており、その仕組みを理解することは、ポストゲノム時代の生命科学分野における先端的研究課題の一つです。モデル微生物である大腸菌はゲノムに約4,700の遺伝子を持っており、それらは約300種類の転写制御因子により制御されていることが分かってきていることから、それら全転写制御因子の機能解明が課題となっています。しかしながら、そのうち約5分の1は未だに機能が全くの不明である機能未知転写因子であり、これらの機能解明を含めたゲノム転写制御機構の全体像の理解が求められています。
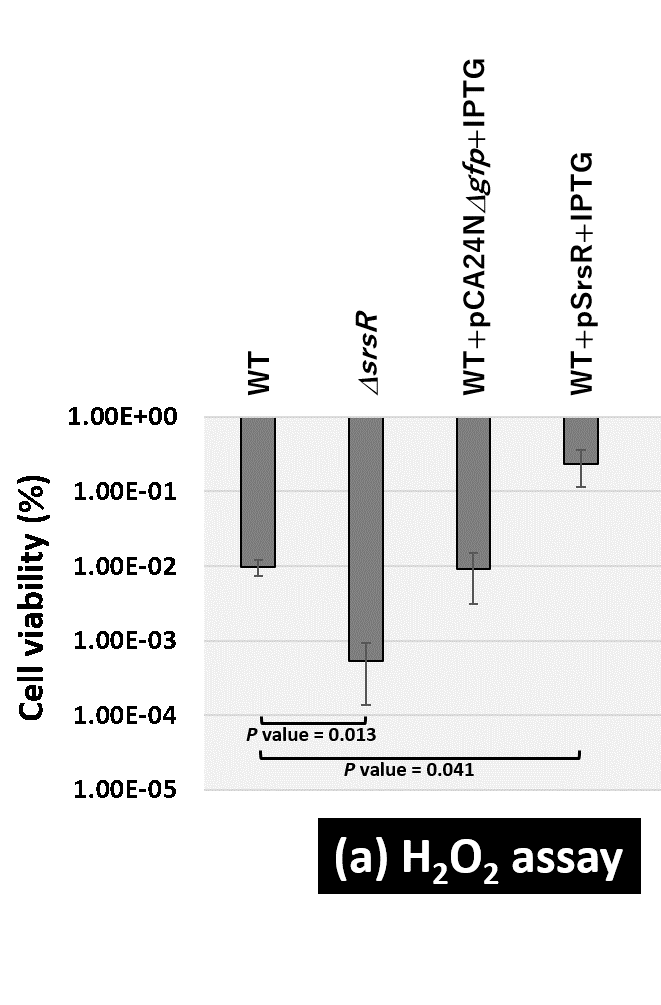
本研究グループは大腸菌をモデル微生物として、一つの生物のゲノム転写制御機構の全体像の理解をめざしています。その一環で、転写因子と推測されている機能未知転写因子YgfIについて、Genomic SELEX法(以下、gSELEX法)を用いてゲノム上の結合領域を解析したところ、約10個の遺伝子群を標的としていることが分かりました(図1)。これらの標的遺伝子群にはバイオフィルム形成や酸化ストレス耐性、多剤耐性、宿主動物腸内での生存などに関与する遺伝子群が含まれていました。次に大腸菌細胞を用いて、YgfIによる制御を観察したところ、YgfIの発現が定常期で上昇し、標的遺伝子群のほとんどは定常期において活性化されていることが分かりました。標的遺伝子群の機能の中でも、バイオフィルム形成や酸化ストレス耐性は、大腸菌の自然環境下や宿主腸内における生存に重要であることが分かっているため、これら表現型へ与えるYgfIの影響を観察しました。その結果、YgfIがバイオフィルム形成を抑制化していること(図2)、および、過酸化水素耐性の獲得に寄与していることが分かりました(図3)。本研究成果から、この機能未知転写因子YgfIをSrsR(a stress-response regulator in stationary phase)と命名することを提案しました。
本研究グループは一つの生物の遺伝子発現制御機構の全体像を理解する目的で、大腸菌をモデル生物としてこれまでに数々の転写制御因子の機能同定に成功してきました。特に、試験管内でゲノム上の直接的な結合配列を網羅的に同定するために独自に開発したgSELEX法を用いた研究戦略により、これまでに大腸菌K-12株の持つ7種類のRNAポリメラーゼシグマ因子のうち6種類、約300種類の転写因子のうち70種類以上の機能同定に成功してきました。特に本研究で解析した機能未知転写因子は、細胞内における発現時期や活性化条件などが不明のため、通常の細胞を用いた機能解析は困難であり、本研究グループのgSELEX法の強みが活かされた研究成果です。今後も、本研究グループによるgSELEX法を用いた機能未知転写因子の機能解析により、微生物が持つ未知の転写制御因子およびその制御機構が明らかとなること、そして、微生物が自然環境下などのストレス条件下における生存のための新たな仕組みが理解されることが期待されます。これらの成果は、微生物のゲノム機能を利用した物質生産や環境浄化、また病原性微生物の理解などの応用分野にも役立つことが期待されます。
明治大学農学部農芸化学科 ゲノム微生物学研究室
図1.本研究グループにより独自に開発されたGenomic SELEX法を用いて同定された機能未知転写因子SrsRの大腸菌ゲノム上の結合領域。結合領域の近傍に位置する遺伝子群が示されている(緑色は遺伝子間領域、橙色は遺伝子上領域を示す)。
図2.大腸菌野生株、srsR欠損株、SrsR過剰発現株、およびコントロール株において形成されたバイオフィルム量の観察結果。
図3.大腸菌野生株、srsR欠損株、コントロール株、SrsR過剰発現株の過酸化水素に対する生残率の観察結果。
研究成果のポイント
- 大腸菌の機能未知転写因子YgfIのゲノム上結合領域および標的プロモーターや標的遺伝子群の網羅的な同定に成功した。標的遺伝子群の多くは、定常期において、バイオフィルム形成や酸化ストレス耐性、多剤耐性、宿主動物腸内での生存に関わる遺伝子であった。
- YgfIが実際に標的遺伝子群の多くを定常期に活性化することが分かった。そして、バイオフィルム形成を抑制し、過酸化水素耐性の獲得に寄与していることを実証した。
- これらの結果から、YgfIをSrsR(a stress-response regulator in stationary phase)と命名することを提案した。本研究成果は、微生物が自然環境下などのストレス条件下で適応するための新たな仕組みの解明・応用に役立つ。
要旨
生物がゲノムに持つ遺伝子を選択的に利用する仕組みを理解することは、ポストゲノム時代の生命科学分野における先端的研究課題の一つです。明治大学の島田友裕准教授と法政大学の石浜明客員教授の研究グループは、大腸菌をモデル微生物として、大腸菌が持つ全ての転写制御因子の機能解明をめざしています。その一環で本研究では、NTT宇宙環境エネルギー研究所の研究グループと共同で、機能未知転写因子YgfIの機能解明を行いました。その結果、YgfIが定常期において、バイオフィルム形成や酸化ストレス耐性、多剤耐性、宿主動物腸内での生存など、微生物が自然環境下におけるストレスに適応するための遺伝子群を活性化していることが示唆されました。さらに、YgfIがバイオフィルム形成の抑制や酸化ストレス耐性の獲得に寄与していることを明らかとしました。これらYgfIによるゲノム転写制御機構の解明から、微生物が自然環境下などのストレス条件下で適応するための新たな仕組みが明らかにされ、YgfIをSrsR(a stress-response regulator in stationary phase)と命名することを提案しました。
本研究は、日本学術振興会による科学研究費基盤C(代表:島田友裕)等の援助により行われました。研究成果は原著論文として、スイスの国際学術誌「International Journal of Molecular Sciences」(電子版)に2022年5月27日付で掲載されました。
1.研究の背景
2.研究内容と成果
3.今後の期待
4.発表論文
<タイトル>
Transcription factor SrsR (YgfI) is a novel regulator for the stress-response genes in stationary phase in Escherichia coli K-12.
<著者名>
Ikki Kobayashi, Kenji Mochizuki, Jun Teramoto, Sousuke Imamura, Kazuhiro Takaya, Akira Ishihama, Tomohiro Shimada
<雑誌名>
International Journal of Molecular Sciences
<DOI>
研究グループ
- 准教授 島田 友裕(しまだ ともひろ)
- 博士研究員 小林 一幾(こばやし いっき)
- 客員教授 石浜 明(いしはま あきら)
- 当時博士研究員 寺本 潤(てらもと じゅん)
- 当時学部生 望月 賢司(もちづき けんじ)
- 特別研究員 今村 壮輔(いまむら そうすけ)
- 主幹研究員 高谷 和宏(たかや かずひろ)
参考図
図1.本研究グループにより独自に開発されたGenomic SELEX法を用いて同定された機能未知転写因子SrsRの大腸菌ゲノム上の結合領域。結合領域の近傍に位置する遺伝子群が示されている(緑色は遺伝子間領域、橙色は遺伝子上領域を示す)。
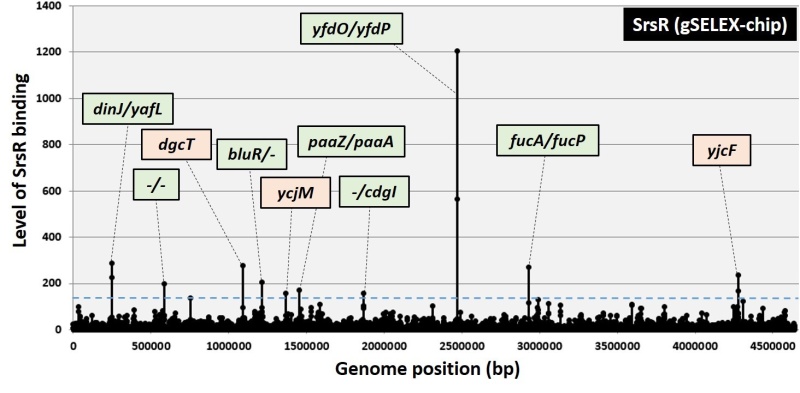
図2.大腸菌野生株、srsR欠損株、SrsR過剰発現株、およびコントロール株において形成されたバイオフィルム量の観察結果。
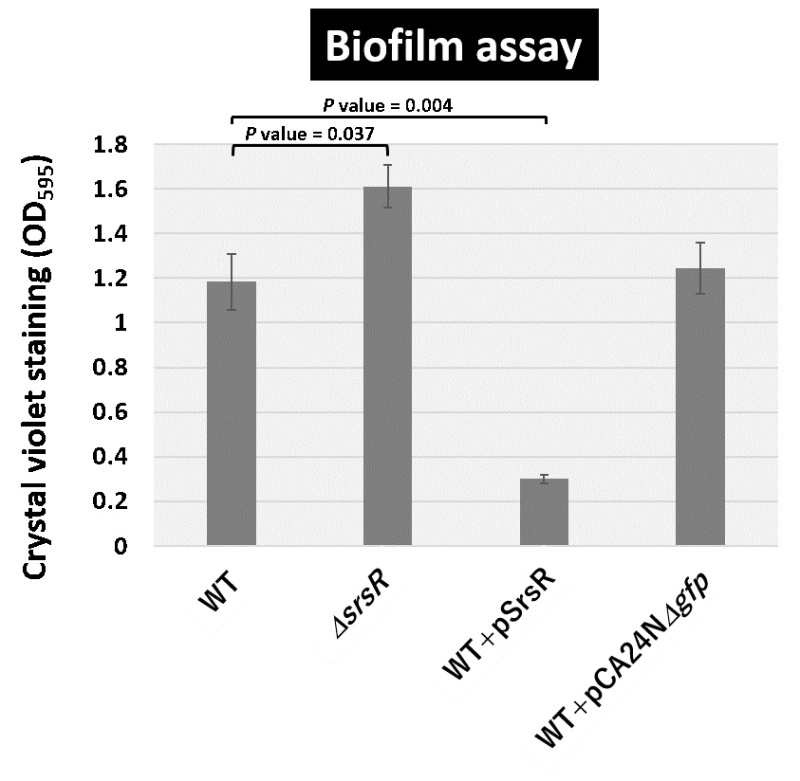
図3.大腸菌野生株、srsR欠損株、コントロール株、SrsR過剰発現株の過酸化水素に対する生残率の観察結果。
- お問い合わせ先
-
<内容に関するお問い合わせ>
明治大学農学部農芸化学科 ゲノム微生物学研究室
准教授 島田 友裕
TEL:044-934-7102 E-mail:tomoshimada@meiji.ac.jp
法政大学マイクロ・ナノテクノロジー研究センター
客員教授 石浜 明
TEL:042-387-5114 E-mail:aishiham@hosei.ac.jp -
<取材に関するお問い合わせ>
明治大学 経営企画部広報課
TEL:03-3296-4082 E-mail:koho@mics.meiji.ac.jp
法政大学 総長室広報課
TEL:03-3264-9240 E-mail:pr@adm.hosei.ac.jp


